Ehemalige Forschungsgruppe Neuroanatomie in der Abteilung Neurophysik – Mikrostrukturelle Konnektivität im Gehirn des Menschen
Hochauflösende Histologie liefert neue Impulse für Magnetresonanztomographie-basierte Traktographie
Neuere Fortschritte in Magnetresonanztomographie (MRT)-basierten biophysikalischen Modellen ermöglichen eine weitaus präzisere Abschätzung der strukturellen Eigenschaften von Faserbahnen der weißen Substanz im menschlichen Gehirn (z.B. relative Myelinisierung, Durchmesser oder Dichte der Nervenzellfortsätze (Axone)). Leider basieren diese Modelle jedoch auf einer starken Vereinfachung der mikrostrukturellen Eigenschaften des Gewebes, z.B. werden Axone als lange parallele Zylinder mit konstantem Durchmesser modelliert. Dies kann zu einer systematischen Über- oder Unterschätzung der ermittelten Parameter führen. Ein weiteres Problem ist der Größenunterschied zwischen einem MRT-Voxel (ca. 500 µm) und der Mikroanatomie des Gewebes (z.B. Axondurchmesser ca. 1µm). Im Gegensatz dazu ermöglicht die hochauflösende licht- und elektronenmikroskopische Analyse von histologischen Schnitten ein weitaus detaillierteres Studium der mikrostrukturellen Eigenschaften der weißen Substanz.
Ziel dieses Projektes ist es, mit hochauflösender Histologie die mikroanatomischen Eigenschaften von Faserbahnen mit genau bekannter anatomischer Orientierung (z.B. Chiasma opticum, Tractus corticospinalis, Corpus callosum) zu definieren. Dies soll zu einer valideren morphologischen Interpretation von strukturellen MRT-Befunden führen.
Wir analysieren Gewebeproben, die bei einer Autopsie (mit vorheriger Einwilligung) von Patienten ohne neurologische oder psychiatrische Erkrankungen gewonnen wurden, fixieren sie mit 3% Paraformaldehyd und 1% Glutaraldehyd und scannen sie mit hochauflösenden strukturellen und diffusionsgewichteten MRT-Sequenzen. Anschließend betten wir das Gewebe in Durcupan-Harz ein und schneiden es mit einem Ultramikrotom mit einer Dicke von 500 nm (Semidünnschnitte) oder 50 nm (Ultradünnschnitte). Die Semidünnschnitte färben wir mit Toluidinblau und digitalisieren sie mit einem hochauflösenden Gewebescanner, die Ultradünnschnitte verwenden wir für Transmissionselektronenmikroskopie.
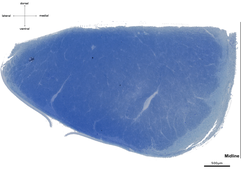
Leider ist die die hochauflösende Lichtmikroskopie von Semidünn- und insbesondere die Elektronenmikroskopie von Ultradünnschnitten durch ein sehr kleines Sichtfeld eingeschränkt. Um dies zu minimieren, verwenden wir spezielle Techniken, die für die Analyse möglichst großer Sichtfelder optimiert wurden. Für eine umfassende Analyse der morphologischen Parameter der weißen Substanz verwenden wir hochauflösende (250 x 250 nm) lichtmikroskopische Aufnahmen von Querschnitten kompletter Faserbahnen, z.B. des Tractus corticospinalis in Höhe der Medulla oblongata (Fläche ca. 10 mm², obere Abbildung).

Teile dieser lichtmikroskopischen Bilder (ca. 100 x 100 µm, untere Abbildung links) segmentieren wir interaktiv in drei strukturelle Kompartimente: (1) axonales Zytoplasma (untere Abbildung, Mitte, grün), (2) Myelinscheiden (rot) und (3) umgebendes Gewebe (blau). Im Anschluss trainieren wir mit diesen Daten einen sog. Deep-Learning Algorithmus. Aus diesen segmentierten Bildern können strukturelle Parameter (z.B. Dichte und Durchmesser der Axone, relative Myelinisierung) errechnet werden. Der trainierte Deep-Learning-Algorithmus kann dann diese mikrostrukturellen Parameter für die kompletten Querschnittsbilder der Faserbahnen berechnen (untere Abbildung, rechts, axonales Zytoplasma in weiß, Myelinscheiden in grau, umgebendes Gewebe in schwarz). Zur Qualitätskontrolle der automatischen Segmentierung der lichtmikroskopischen Bilder verwenden wir benachbarte Ultradünnschnitte mit elektronenmikroskopischer Auflösung.
In Kombination mit den MRT-Daten der gleichen Gewebeproben erwarten wir von diesen hochauflösenden histologischen Messungen ein besseres Verständnis der Mikrostruktur der weißen Substanz und vor allem der morphologischen Grundlagen der MRT-basierten biophysikalischen Modelle.

